- Giới thiệu Hệ vi khuẩn đường tiêu hóa (GIT) là thành phần chính trong sức khỏe, dinh dưỡng, điều hòa sinh lý, phát triển ruột và tăng trưởng của gia cầm. Trong hai thập kỷ qua, việc phát triển và chi phí thấp hơn của công nghệ giải trình tự thế hệ mới (NGS) đã dẫn đến sự gia tăng nhanh chóng các công bố về hệ vi sinh vật gia cầm. Giải trình tự dựa trên amplicon gen 16S ribosomal RNA (rRNA) vẫn là phương pháp NGS được sử dụng rộng rãi nhất trong các nghiên cứu hệ vi sinh vật gia cầm. Tuy nhiên, hiện tại không có giao thức chuẩn hóa cho việc phân tích hồ sơ gen 16S rRNA và lưu trữ dữ liệu cho các nghiên cứu hệ vi sinh vật gia cầm. Các biến thể trong lưu trữ và phân lập DNA, thiết kế mồi (primer), và chuẩn bị thư viện đều có thể gây ra sai lệch, ảnh hưởng đến cấu trúc cộng đồng và phân tích quần thể vi sinh vật. Điều này khiến kết quả nghiên cứu hệ vi sinh vật gia cầm thường khó tái lập và so sánh giữa các nghiên cứu.
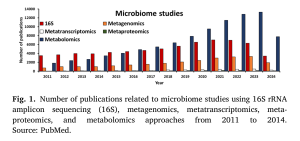
- Tổng quan về giải trình tự khuếch đại gen 16S rRNA Hệ vi sinh vật đã được nghiên cứu bằng nhiều kỹ thuật sinh hóa, vi sinh, miễn dịch và sinh học phân tử. Trong số các phương pháp phân tích trình tự thế hệ mới (NGS), giải trình tự amplicon (giải trình tự gen 16S rRNA) và metagenomics shotgun là phổ biến nhất. Trong nghiên cứu hệ vi sinh vật gia cầm, giải trình tự amplicon là phương pháp được sử dụng rộng rãi, đặc biệt với các nền tảng Illumina HiSeq và MiSeq. Một tìm kiếm trên PubMed cho thấy giải trình tự khuếch đại gen 16S rRNA là phương pháp phổ biến nhất để lập hồ sơ phân loại trong 13 năm qua.
Gen 16S rRNA của vi khuẩn, dài khoảng 1.500 bp, được bảo tồn cao giữa các loài vi khuẩn khác nhau, là mục tiêu lý tưởng để nhận dạng vi khuẩn. Các vùng bảo tồn cao cho phép thiết kế các mồi phổ rộng, trong khi các vùng siêu biến đổi cho phép phân biệt phân loại. Tuy nhiên, sự bảo tồn trình tự trong các vùng siêu biến đổi này có thể hạn chế khả năng phân biệt cấp độ loài đối với các loài có quan hệ gần gũi. Vi khuẩn có thể chứa nhiều bản sao của gen 16S rRNA, điều này có thể làm sai lệch ước tính về sự phong phú tương đối của chúng, khiến vi khuẩn có nhiều bản sao gen 16S rRNA xuất hiện nhiều hơn thực tế5. Ví dụ, Clostridium perfringens có trung bình 10 bản sao gen 16S rRNA trên mỗi bộ gen, trong khi Escherichia coli có 7 bản sao, và Helicobacter và Campylobacter chỉ có 2 và 3 bản sao tương ứng.
Gen 16S rRNA bao gồm cả vùng bảo tồn và vùng biến đổi. Các vùng bảo tồn rất giống nhau giữa các loài vi khuẩn khác nhau, cung cấp mục tiêu ổn định cho việc gắn mồi trong quá trình khuếch đại PCR6. Ngược lại, các vùng biến đổi thể hiện sự đa dạng trình tự đáng kể, cho phép phân biệt và nhận dạng các đơn vị phân loại vi khuẩn cụ thể. Có chín vùng siêu biến đổi, được gọi là V1-V9, xen kẽ giữa các vùng bảo tồn. Bằng cách nhắm mục tiêu các vùng biến đổi này, thành phần của một cộng đồng vi khuẩn phức tạp có thể được tiết lộ. Phương pháp gen 16S rRNA cho phép khuếch đại và giải trình tự một hoặc nhiều vùng siêu biến đổi của gen.
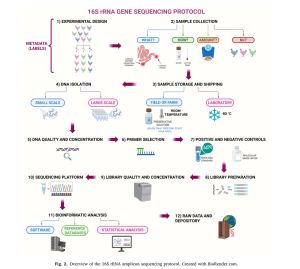
- Thiết kế thực nghiệm Thiết kế một nghiên cứu hệ vi sinh vật gia cầm liên quan đến nhiều cân nhắc quan trọng để đảm bảo tính hợp lệ và độ tin cậy của kết quả. Các biến số như tuổi, giống, vùng GIT/loại mẫu, yếu tố mẹ, giới tính, thức ăn, hệ thống và điều kiện chuồng trại, điều kiện vệ sinh/làm sạch, tình trạng tiêm phòng, sử dụng kháng sinh, nhiệt độ, chất độn chuồng và môi trường đều ảnh hưởng đáng kể đến cấu trúc và chức năng của hệ vi sinh vật. Việc xác định kích thước mẫu thích hợp cũng là một khía cạnh quan trọng của thiết kế nghiên cứu.
- Thu thập mẫu Thu thập mẫu là giai đoạn đầu tiên của một nghiên cứu hệ vi sinh vật. Cách thức thu thập mẫu sẽ ảnh hưởng đến mọi giai đoạn tiếp theo của quy trình nghiên cứu hệ vi sinh vật, đóng vai trò quyết định đến chất lượng và tính hợp lệ của bộ dữ liệu hệ vi sinh vật.
Giải phẫu và chức năng của GIT gia cầm – tầm quan trọng trong thu thập mẫu:
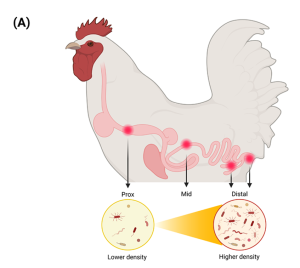
Vấn đề sinh khối vi sinh vật: GIT gia cầm bao gồm nhiều vùng riêng biệt, mỗi vùng có chức năng sinh học riêng. Sinh khối vi sinh vật (cụ thể là vi khuẩn) tăng lên dọc theo GIT gia cầm. Tương tự, có một gradient mật độ vi sinh vật theo mặt cắt ngang từ niêm mạc (ít dày đặc nhất) đến dịch lòng ruột (dày đặc nhất) của mỗi vùng GIT.
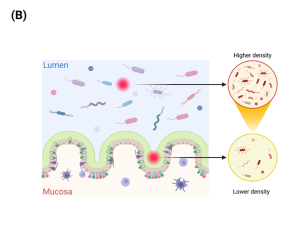
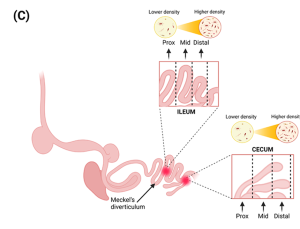
Độ đặc của dịch lòng ruột: Dịch lòng ruột của mỗi vùng giải phẫu của GIT có những cân nhắc về độ đặc vật lý riêng biệt cho việc thu thập mẫu.
Từ phần nào của vùng nào trong GIT?: Điều quan trọng là phải nhấn mạnh rằng chiều dài của mỗi phần GIT cần được cân nhắc khi thu thập mẫu. Mẫu được thu thập từ phần gần, giữa hay xa của vùng GIT sẽ ảnh hưởng đến các đơn vị phân loại vi khuẩn được xác định.

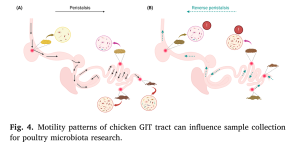
Nhu động ngược: Hiện tượng nhu động ngược, mô tả sự di chuyển ngược (từ lỗ huyệt về phía diều) của dịch lòng đường tiêu hóa, là duy nhất ở gia cầm. Mặc dù nhu động ngược có thể di chuyển các mầm bệnh vi khuẩn tiềm năng giữa các vùng GIT, nhưng tác động của nó đối với sự phân bố vùng của các cộng đồng vi sinh vật chưa được nghiên cứu chính thức.
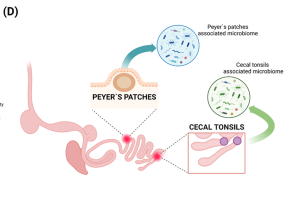
Ô nhiễm trong quá trình thu thập mẫu: Vấn đề ô nhiễm luôn là một mối quan tâm hiện hữu trong quá trình thu thập mẫu. Các điều kiện trang trại có thể ảnh hưởng đến việc thu thập mẫu, ví dụ như bụi trong chuồng gia cầm có thể chứa nhiều đơn vị phân loại vi khuẩn tương tự như được tìm thấy trong dịch manh tràng. Điều quan trọng là phải đánh giá các công cụ và kỹ thuật thu thập mẫu, và nhà nghiên cứu có thể là một nguồn biến đổi trong việc thu thập mẫu.
Thu thập mẫu xâm lấn so với không xâm lấn: Việc thu thập dịch lòng hoặc mẫu niêm mạc GIT yêu cầu phải euthanize chim. Các phương pháp thu thập mẫu không xâm lấn cho hệ vi sinh vật GIT ở gia cầm có thể được thực hiện mà không ảnh hưởng tiêu cực đến chim, bao gồm: phân manh tràng, mẫu tất ủng và tăm bông lỗ huyệt. Các phương pháp không xâm lấn chỉ nắm bắt được một phần sự phức tạp của hệ vi sinh vật so với các phương pháp xâm lấn.
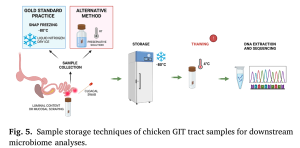
- Lưu trữ mẫu Giai đoạn thứ hai trong nghiên cứu hệ vi sinh vật gia cầm là lưu trữ mẫu. Tiêu chuẩn vàng để bảo quản mẫu vi sinh vật vẫn là đông lạnh tức thì (snap freezing), sau đó là lưu trữ ở -80°C1920. Tất cả các phương pháp lưu trữ đều có thể ảnh hưởng, ở các mức độ khác nhau, đến thành phần vi sinh vật được xác định trong nghiên cứu giải trình tự khuếch đại gen 16S rRNA.
- Phân lập, định lượng và chất lượng DNA Chiết tách DNA là một bước quan trọng trong các nghiên cứu hệ vi sinh vật gia cầm, vì nó ảnh hưởng đến độ chính xác và khả năng tái lập của các hồ sơ vi sinh vật.
Việc lựa chọn phương pháp chiết tách DNA có thể dẫn đến sự biến đổi đáng kể trong sự biểu hiện của các đơn vị phân loại vi khuẩn. Các bộ chiết tách thương mại khác nhau sử dụng các giao thức khác nhau để phân giải tế bào vi khuẩn, chẳng hạn như tiêu hóa bằng enzyme và hóa chất, và phá vỡ cơ học bằng phương pháp bead-beating. Phương pháp phá vỡ cơ học, đặc biệt là bead-beating sau tiêu hóa hóa học, được khuyến nghị để phân giải hiệu quả vi khuẩn Gram dương. Ngược lại, việc thiếu bước bead-beating trong giao thức chiết tách có thể dẫn đến loại trừ hoàn toàn một số vi khuẩn Gram dương. Tuy nhiên, bead-beating quá mức có thể dẫn đến cắt đứt DNA, làm cho DNA bị phân mảnh và có thể ảnh hưởng đến giải trình tự tiếp theo.
- Kiểm soát dương tính và âm tính Thực hành khoa học tốt cần bao gồm các kiểm soát trong quy trình thực nghiệm, và nghiên cứu hệ vi sinh vật cũng không ngoại lệ. Kiểm soát dương tính và âm tính đóng vai trò quan trọng trong việc giải thích, tính hợp lệ, khả năng tái lập và khả năng tái sử dụng dữ liệu hệ vi sinh vật.
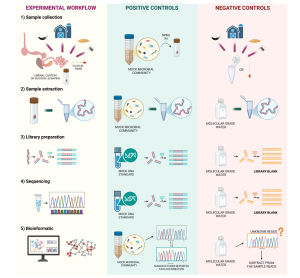
Kiểm soát dương tính: Kiểm soát dương tính thường đề cập đến các tiêu chuẩn tế bào vi khuẩn nguyên vẹn (tức là các cộng đồng vi sinh vật mô phỏng) hoặc các tiêu chuẩn DNA vi khuẩn đã chiết tách (tức là các mẫu DNA mô phỏng). Chúng được sử dụng để đánh giá hiệu suất và/hoặc sai lệch ở mỗi giai đoạn của quy trình thực nghiệm.
Kiểm soát âm tính: Kiểm soát âm tính chủ yếu liên quan đến vấn đề ô nhiễm . Tương tự như kiểm soát dương tính, một kiểm soát âm tính có thể được thêm vào ở mỗi giai đoạn của nghiên cứu hệ vi sinh vật và nên được thực hiện qua tất cả các bước tiếp theo.
- Lựa chọn mồi Có 9 vùng siêu biến đổi trong gen 16S rRNA của vi khuẩn được bao quanh bởi các vùng bảo tồn cao. Các vùng siêu biến đổi được sử dụng để tính toán các mối quan hệ tiến hóa và sự tương đồng giữa các loài, trong khi các vùng bảo tồn thường được sử dụng để thiết kế các mồi PCR. Các nghiên cứu đã chỉ ra rằng việc lựa chọn bộ mồi gen 16S rRNA có thể ảnh hưởng đến phân tích hệ vi sinh vật và việc giải thích kết quả ở gia cầm.
- Nền tảng giải trình tự Trong những năm qua, các nền tảng được sử dụng rộng rãi như Roche 454 GSFLX+, Ion Torrent Personal Genome Machine (PGM), và Pacific Biosciences (PacBio) Single Molecule Real-Time (SMRT) đã được thay thế bằng Oxford Nanopore Technologies (ONT) và Illumina MiSeq, với Illumina MiSeq là nền tảng được sử dụng phổ biến nhất trong các nghiên cứu gần đây.
- Độ sâu giải trình tự Độ sâu giải trình tự nhất quán là một cân nhắc quan trọng để tránh việc xác định các đơn vị phân loại vi khuẩn không đồng đều giữa các mẫu. Hiện tại, không có hướng dẫn về độ sâu giải trình tự cho các vùng khác nhau của đường tiêu hóa gia cầm.
- Lựa chọn cơ sở dữ liệu Hồ sơ phân loại của các mẫu giải trình tự amplicon 16S rRNA phụ thuộc vào các cơ sở dữ liệu công khai. Trong số ba cơ sở dữ liệu được sử dụng phổ biến nhất, SILVA là lớn nhất dựa trên phân loại gen 16S rRNA, tiếp theo là RDP và Greengenes. Việc lựa chọn cơ sở dữ liệu có thể ảnh hưởng đến kết quả và việc giải thích kết quả.
- Tin sinh học Các công cụ tin sinh học để phân tích dữ liệu giải trình tự là quan trọng để loại bỏ các tạo tác và chimeras có thể làm sai lệch kết quả giải trình tự. Sự phát triển của giải trình tự thế hệ mới đã thúc đẩy sự phát triển nhanh chóng của các quy trình tin sinh học để phân tích dữ liệu hệ vi sinh vật gen 16S rRNA; các công cụ được sử dụng rộng rãi nhất là Quantitative Insights Into Microbial Ecology (QIIME), UPARSE và Mothur.
QIIME2: QIIME là một quy trình mã nguồn mở được phát triển để phân tích dữ liệu cộng đồng thông lượng cao3. Nó tạo ra các biến thể trình tự amplicon (ASV), đại diện cho các trình tự duy nhất với độ chính xác một nucleotide, mang lại độ phân giải phân loại tốt hơn và giảm thiểu ảnh hưởng của lỗi giải trình tự so với các đơn vị phân loại hoạt động (OTU).
UPARSE: Quy trình UPARSE được phát triển để tạo ra các OTU thông lượng cao từ nhiều loại dữ liệu amplicon.
Mothur: Mothur được giới thiệu lần đầu tiên vào năm 2009 như một nền tảng phần mềm mã nguồn mở duy nhất bao gồm nhiều thuật toán từ các công cụ trước đó.
- Metadata Metadata là “dữ liệu về dữ liệu” đại diện cho thông tin liên quan đến dữ liệu thực nghiệm và cung cấp thông tin toàn diện về nguồn gốc mẫu. Mặc dù metadata rất quan trọng để hiểu và giải thích kết quả, việc thu thập metadata thường kém chuẩn hóa. Metadata là rất quan trọng trong các nghiên cứu hệ vi sinh vật gia cầm vì nó cho phép các nhà nghiên cứu tính đến và phân tích nhiều biến số ảnh hưởng đáng kể đến các cộng đồng vi sinh vật.
- Phân tích thống kê Dữ liệu hệ vi sinh vật được tạo ra bằng giải trình tự khuếch đại 16S rRNA có các thuộc tính độc đáo, ví dụ như sự lạm phát bằng không (zero inflation) và bản chất là dữ liệu thành phần (compositional), có nghĩa là dữ liệu đại diện cho sự phong phú tương đối chứ không phải số lượng tuyệt đối.
Đa dạng Alpha và Beta: Các câu hỏi nghiên cứu thường tập trung vào ảnh hưởng của sự đa dạng. Đa dạng Alpha đề cập đến sự biến đổi trong một mẫu duy nhất, trong khi đa dạng Beta đo lường sự biến đổi giữa các mẫu.
Phân tích sự phong phú khác biệt (DA): Loại kiểm định này nhằm tìm ra sự khác biệt về phân loại giữa hai nhóm. Các kỹ thuật như ANCOM-BC2 được khuyến nghị cao, đặc biệt phù hợp cho việc so sánh nhiều nhóm và đo lường lặp lại.
Chuẩn hóa dữ liệu: Chuẩn hóa dữ liệu là một bước quan trọng trong phân tích hệ vi sinh vật để giải quyết sai lệch thành phần và sự biến đổi trong độ sâu giải trình tự giữa các mẫu. Các cách tiếp cận được áp dụng phổ biến bao gồm: rarefaction, biến đổi dữ liệu thành phần (đặc biệt là biến đổi log-ratio tập trung CLR), và cân bằng tổng lũy tích (CSS).
Dự đoán chức năng chuyển hóa: Một số nhà nghiên cứu sử dụng giải trình tự khuếch đại 16S rRNA để dự đoán các chức năng chuyển hóa, thông qua công cụ PICRUSt2.
- Tích hợp dữ liệu Tích hợp dữ liệu đã trở thành một thành phần quan trọng trong việc nâng cao hiểu biết của chúng ta về hệ vi sinh vật gia cầm. Bằng cách kết hợp các loại dữ liệu đa dạng bao gồm genomics, transcriptomics, proteomics, metabolomics, thành phần thức ăn, phản ứng miễn dịch và điều kiện môi trường, các nhà nghiên cứu có thể khám phá các tương tác phức tạp trong hệ vi sinh vật.
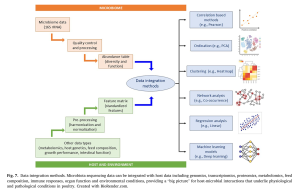
Phương pháp tương quan: Các phương pháp dựa trên tương quan là quan trọng trong các nghiên cứu hệ vi sinh vật gia cầm, giúp hiểu các mối quan hệ giữa các cộng đồng vi sinh vật và các yếu tố vật chủ.
Phương pháp sắp xếp và phân cụm: Phương pháp sắp xếp và phân cụm là công cụ thiết yếu để xử lý dữ liệu hệ vi sinh vật có chiều cao và xác định các mẫu trong các cộng đồng vi sinh vật phức tạp. Phân tích thành phần chính (PCA) và Phân tích tọa độ chính (PCoA) là các kỹ thuật phổ biến được sử dụng để giảm chiều dữ liệu lớn và trực quan hóa sự khác biệt tổng thể giữa các mẫu. Phân tích tương ứng chính tắc (CCA) được sử dụng để khám phá mối quan hệ giữa các cộng đồng vi sinh vật và gradient môi trường.
Phương pháp mạng lưới: Các phương pháp mạng lưới cung cấp một khuôn khổ mạnh mẽ để trực quan hóa và phân tích các tương tác phức tạp trong các cộng đồng vi sinh vật và giữa các vi sinh vật với các yếu tố vật chủ.
Phương pháp hồi quy: Kỹ thuật hồi quy là rất quan trọng trong nghiên cứu hệ vi sinh vật để mô hình hóa mối quan hệ giữa sự phong phú của vi sinh vật và các kết quả hoặc yếu tố dự đoán khác nhau.
Mô hình học máy: Gần đây, ngày càng có nhiều sự quan tâm đến việc sử dụng các mô hình học máy trong nghiên cứu hệ vi sinh vật để dự đoán kết quả sức khỏe và tích hợp các bộ dữ liệu phức tạp.
- Kho dữ liệu thô Các kho dữ liệu công cộng là một nguồn tài nguyên vô giá để lưu trữ dài hạn và duy trì khả năng truy cập vào dữ liệu giải trình tự và siêu dữ liệu liên quan.
Mặc dù không có cơ sở dữ liệu trình tự DNA/RNA chuyên biệt cho gia cầm, nhưng các kho trình tự hiện có sẵn dễ dàng chấp nhận các bộ dữ liệu được tạo ra từ các loài gia cầm. Có ba cơ sở dữ liệu trình tự DNA/RNA thành viên của Hợp tác xã Cơ sở dữ liệu Trình tự Nucleotide Quốc tế (INSDC): Thư viện Lưu trữ Trình tự Đọc Quốc gia (NCBI-SRA), Lưu trữ Nucleotide Châu Âu (ENA) và Ngân hàng Dữ liệu DNA Nhật Bản (DDJ-SRA).
- Kế hoạch tương lai và kết luận Xét các chiến lược sử dụng hệ vi sinh vật hứa hẹn sẽ cách mạng hóa sản xuất gia cầm, điều quan trọng là phải hiểu các yếu tố đặc thù của gia cầm có thể ảnh hưởng đến thiết kế thực nghiệm hệ vi sinh vật để cuối cùng ảnh hưởng đến chất lượng và khả năng tái lập dữ liệu. Hiện tại, ít sự chú ý được dành cho câu hỏi về cách sinh lý gia cầm và môi trường sản xuất có thể đưa ra các cân nhắc sinh học và hậu cần để định hình các phương pháp được sử dụng ở mỗi giai đoạn thực nghiệm của một nghiên cứu hệ vi sinh vật gia cầm55. Do đó, mục tiêu của bài đánh giá này là hai mặt: đầu tiên, xác định toàn diện các yếu tố đặc thù của gia cầm có thể định hình từng giai đoạn của thiết kế thực nghiệm hệ vi sinh vật; và thứ hai, thiết lập một lý do dựa trên bằng chứng cho sự cần thiết của một phương pháp luận thực hành tốt nhất cho các nghiên cứu giải trình tự khuếch đại gen 16S rRNA hệ vi sinh vật gia cầm55. Câu hỏi đặt ra trong tiêu đề của bài đánh giá này, cụ thể là ‘Chúng ta có cần một giao thức phân tích nghiên cứu giải trình tự khuếch đại gen 16S rRNA chuẩn hóa trong nghiên cứu hệ vi sinh vật gia cầm không?’, giờ đây có thể được trả lời một cách dứt khoát là, ‘CÓ’.
LOBI Vietnam là công ty tiên phong trong lĩnh vực Đọc trình tự gen thế hệ mới NGS (Next Generation Sequencing) và Phân tích Tin sinh học. Liên hệ hotline/Zalo 092.510.8899 để biết thêm chi tiết.


