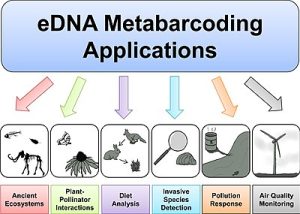
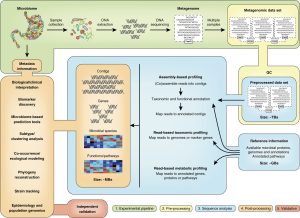
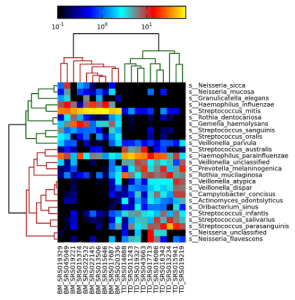
Metagenomics là một lĩnh vực khoa học nghiên cứu đa dạng sinh học bằng cách phân tích và hiểu các cộng đồng vi sinh vật (microbiome) trong môi trường tự nhiên hoặc trong các mẫu sinh học, như đất, nước, thực vật, động vật, con người và các môi trường khác. Nó liên quan đến việc phân tích DNA hoặc RNA của các vi khuẩn, virus, nấm và các sinh vật khác trong một mẫu, bao gồm cả những sinh vật chưa được mô tả trước đó.
Chuyên mục blog Metagenomics này cung cấp một loạt các bài viết liên quan tới Metagenomics, từ khái niệm cho tới các công cụ Tin sinh học được sử dụng để phân tích, cũng như các ưu nhược điểm của chúng.
Phát hiện Sớm Virus Sởi trong Nước thải: Góc nhìn từ Tin sinh học
Giới thiệu Bệnh Sởi: Mối Đe Dọa Sức Khỏe Toàn Cầu Bệnh sởi, dù đã có vắc-xin hiệu quả, vẫn là một thách thức lớn đối với sức khỏe cộng đồng. Các đợt bùng phát bệnh sởi tiếp tục xảy ra do sự do dự tiêm vắc-xin, khả năng miễn...
Th5
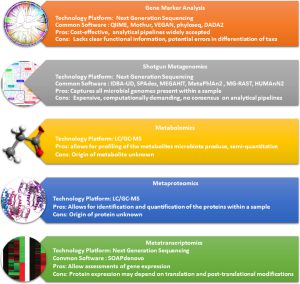
Một số công cụ tin sinh (tools) phân tích hệ vi sinh vật
Phân tích hệ vi sinh vật (microbiome) đã trở thành một lĩnh vực nghiên cứu quan trọng nhờ sự phát triển của các phương pháp và công cụ hiện đại. Bài viết dưới đây sẽ đưa ra một số phương pháp được sử dụng phổ biến trong nghiên cứu. Phân...
Th3
Hệ vi sinh vật đường ruột và bệnh tiểu đường
Hệ vi sinh vật, bao gồm vi khuẩn, nấm và vi rút, đóng một vai trò quan trọng trong nhiều chức năng của cơ thể con người, bao gồm chuyển hóa, miễn dịch và thậm chí cả sức khỏe tâm thần. Sự mất cân bằng trong hệ vi sinh vật...
Th11
Metagenomics: Phát Hiện Mầm Bệnh Của Mẫu Huyết Tương Từ Bệnh Nhân Bị Nhiễm Trùng Huyết ở Uganda
Trong bài viết này, chúng tôi tóm lược nội dung nghiên cứu được công bố năm 2023 bởi Grundy và cộng sự trên tạp chí Microbiology Spectrum (DOI). Bài báo này trình bày kết quả của một nghiên cứu được thực hiện tại Uganda, nhằm mục đích sử dụng giải...
Th11
Phân Tích Metagenomics Trong Đất Trồng Lúa: Một Cái Nhìn Chi Tiết
Phân tích metagenomics đã trở thành một công cụ quan trọng trong việc nghiên cứu hệ sinh thái đất trồng lúa, mang đến những hiểu biết sâu sắc về sự đa dạng, chức năng và tương tác phức tạp của cộng đồng vi sinh vật trong môi trường đặc biệt...
Th11
Ứng dụng của Metagenomics trong nghiên cứu về đất
Metagenomics là gì? Metagenomics là nghiên cứu vật chất di truyền thu được trực tiếp từ các mẫu môi trường. Thay vì nuôi cấy các vi sinh vật riêng lẻ, metagenomics cho phép các nhà nghiên cứu nghiên cứu toàn bộ cộng đồng vi sinh vật trong môi trường sống...
Th11
Cơ sở dữ liệu Silva ribosomal RNA database
Giới thiệu về Silva ribosomal RNA database Cơ sở dữ liệu RNA ribosomal Silva là một nguồn tài nguyên quan trọng trong lĩnh vực sinh thái vi sinh vật và phân loại vi sinh vật. Nó cung cấp dữ liệu chuỗi RNA ribosomal được thu thập từ các hệ vi...
Th6
Metabarcoding là gì?
Metabarcoding là gì? Metabarcoding là một phương pháp khoa học được sử dụng để nghiên cứu đa dạng sinh học trong một mẫu môi trường bằng cách sử dụng chuỗi gen. Phương pháp này thường được áp dụng trong lĩnh vực sinh thái học môi trường và đa dạng sinh...
Th5
Best practice trong lấy mẫu metagenomics
Việc lấy mẫu là một bước quan trọng trong phân tích metagenomics, và có thể gặp phải một số vấn đề sau đây: Sự đa dạng của mẫu: Mẫu metagenomics có thể bao gồm nhiều loại vi sinh vật khác nhau, bao gồm cả vi khuẩn, nấm, vi rút và...
Th4
MetaPhlAn – công cụ phân tích shotgun metagenomics
MetaPhlAn được phát triển bởi Huttenhower Lab tại Đại học Harvard và miễn phí cho việc sử dụng học thuật và phi thương mại. MetaPhlAn là một công cụ Tin sinh học để phân tích hỗn hợp vi sinh vật (Bacteria, Archaea và Eukaryotes) từ dữ liệu đọc trình tự...
Th3