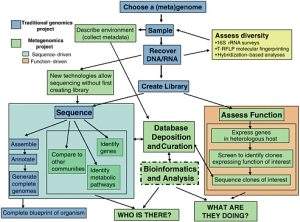
Metagenomics là một lĩnh vực khoa học nghiên cứu đa dạng sinh học bằng cách phân tích và hiểu các cộng đồng vi sinh vật (microbiome) trong môi trường tự nhiên hoặc trong các mẫu sinh học, như đất, nước, thực vật, động vật, con người và các môi trường khác. Nó liên quan đến việc phân tích DNA hoặc RNA của các vi khuẩn, virus, nấm và các sinh vật khác trong một mẫu, bao gồm cả những sinh vật chưa được mô tả trước đó.
Chuyên mục blog Metagenomics này cung cấp một loạt các bài viết liên quan tới Metagenomics, từ khái niệm cho tới các công cụ Tin sinh học được sử dụng để phân tích, cũng như các ưu nhược điểm của chúng.
Shotgun metagenomics: hiểu rõ về hệ vi sinh vật
Giới thiệu Các cộng đồng vi sinh vật là những hệ sinh thái phức tạp và đa dạng đóng vai trò quan trọng trong nhiều khía cạnh của cuộc sống, từ việc duy trì sức khỏe cho đến đẩy mạnh quá trình sinh hóa địa hóa trong môi trường. Việc...
Th3
Phân tích 16S metagenomics với MOTHUR
Mothur là một phần mềm Tin sinh học miễn phí và mã nguồn mở được sử dụng trong phân tích dữ liệu 16S metagenomics. Nó cung cấp một loạt các công cụ và chức năng để thực hiện các bước khác nhau trong việc phân tích dữ liệu 16S metagenomics,...
Th2
Phân tích metagenomics với QIIME
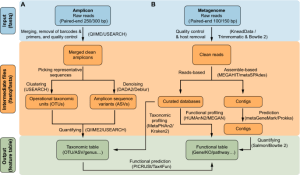
Quy trình phân tích QIIME QIIME (Quantitative Insights Into Microbial Ecology) là một pipeline Tin sinh học phổ biến để phân tích dữ liệu metagenomic. Đây là một phần mềm miễn phí và mã nguồn mở cung cấp một bộ công cụ toàn diện để thực hiện kiểm soát chất...
Th2
Pipeline phân tích 16S/ITS metagenomics phổ biến
Có nhiều pipeline được sử dụng để phân tích dữ liệu 16S/ITS metagenomics, tùy thuộc vào mục đích và tính năng của từng pipeline. Dưới đây là một số pipeline phổ biến được sử dụng nhiều trong phân tích 16S/ITS metagenomics: QIIME (Quantitative Insights Into Microbial Ecology): QIIME là một...
Th2
Best practices trong phân tích 16S/ITS metagenomics
Kiểm soát chất lượng dữ liệu đọc trình tự: Kiểm soát chất lượng là một bước cần thiết trong phân tích dữ liệu metagenomic 16S. Nó bao gồm lọc các read có chất lượng lượng thấp, loại bỏ các read có độ dài ngắn, cắt các chuỗi adapter và loại...
Th2
Những ứng dụng nổi trội của 16S amplicon sequencing?
16S amplicon sequencing là một công cụ quan trọng trong vi sinh học và đã được sử dụng rộng rãi trong nhiều lĩnh vực khác nhau. Sau đây là những ứng dụng nổi trội của metagenomic 16S: Nghiên cứu vi sinh vật đồng sinh: 16S amplicon sequencing được sử dụng...
Th2
Xu hướng nghiên cứu và ứng dụng 16S amplicon sequencing trong những năm qua?
Trong những năm gần đây, nghiên cứu và ứng dụng của 16S amplicon sequencing đã phát triển mạnh mẽ và có nhiều xu hướng như sau: Sự phát triển của công nghệ NGS (Next-Generation Sequencing) đã giúp tăng đáng kể độ phân giải và khả năng xác định các loài...
Th2
16S amplicon sequencing là gì?
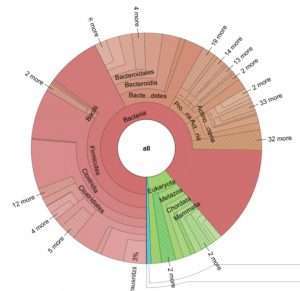
16S amplicon sequencing còn được gọi là Metagenomic 16S, 16S rRNA gene sequencing hoặc 16S ribosomal RNA sequencing. Nó là một phương pháp phổ biến trong việc phân tích đa dạng vi khuẩn trong các mẫu sinh thái học bằng cách phân tích các đoạn gen 16S rRNA có mặt...
Th2
Tổng kết Webinar 01 | Metagenomics: xu thế, thuận lợi và khó khăn
Ngày 16 tháng 4 năm 2022, LOBI Việt Nam đã tổ chức thành công Webinar 01 với chủ đề Metagenomics: xu thế, thuận lợi và khó khăn do NCS. Phạm Quang Huy trình bày. Webinar đã thu hút được hơn 200 lượt đăng ký tham dự, với đối tượng người...
Th4
Metagenomics: Chọn V1V2 hay V3V4?
Sau khi đăng bài Phân tích metagenomics: 16S, 18S, ITS hay shotgun?, chúng tôi tiếp tục nhận được câu hỏi: Nên chọn V1V2 hay V3V4 cho nghiên cứu metagenomics? Như các bạn đã biết, 16S rRNA được sử dụng để phân tích đa dạng của các loài vi khuẩn hoặc...
Th3