MỤC LỤC BÀI VIẾT
Tổng quan và đặt vấn đề
- Dự đoán bộ gen (Genomic Prediction – GP) là một phương pháp hiệu quả để dự đoán các tính trạng kiểu hình từ thông tin kiểu gen, giúp đẩy nhanh quá trình cải thiện tính trạng trong chọn giống cây trồng.
- Các phương pháp GP truyền thống chủ yếu dựa trên các mô hình hỗn hợp tuyến tính như GBLUP (Genomic Best Linear Unbiased Prediction) và các phương pháp học máy thông thường như SVR (Support Vector Regression). Các phương pháp truyền thống này có hạn chế trong việc xử lý dữ liệu chiều cao (high-dimensional) và các mối quan hệ phi tuyến tính.
- Các phương pháp học sâu (deep learning – DL) đã được áp dụng cho GP gần đây để khắc phục những hạn chế này. Tuy nhiên, các mô hình DL dựa trên CNN (Convolutional Neural Network) hiện tại thường mã hóa các điểm đa hình đơn nucleotide (SNP) một cách đơn giản (ví dụ: 0, 1, 2), điều này mâu thuẫn với tính cục bộ (locality) của CNN và bỏ qua thông tin sinh học quan trọng. Chúng cũng gặp khó khăn trong việc tích hợp hiệu quả dữ liệu đa-omics.
- Để giải quyết các vấn đề trên, các tác giả đã đề xuất mô hình iADEP, một mô hình dựa trên học sâu tích hợp kiến thức sinh học tiên nghiệm (biological prior knowledge) và cho phép kết hợp dữ liệu đa-omics tùy chọn.
Mô hình iADEP (Integrated Additive, Dominant, and Epistatic Prediction)
- iADEP chủ yếu gồm ba phần: nhúng SNP (SNP embedding), bộ mã hóa cục bộ-bộ giải mã toàn cục (local encoder-global decoder), và đa tầng Perceptron (MLP – multilayer perceptrons)
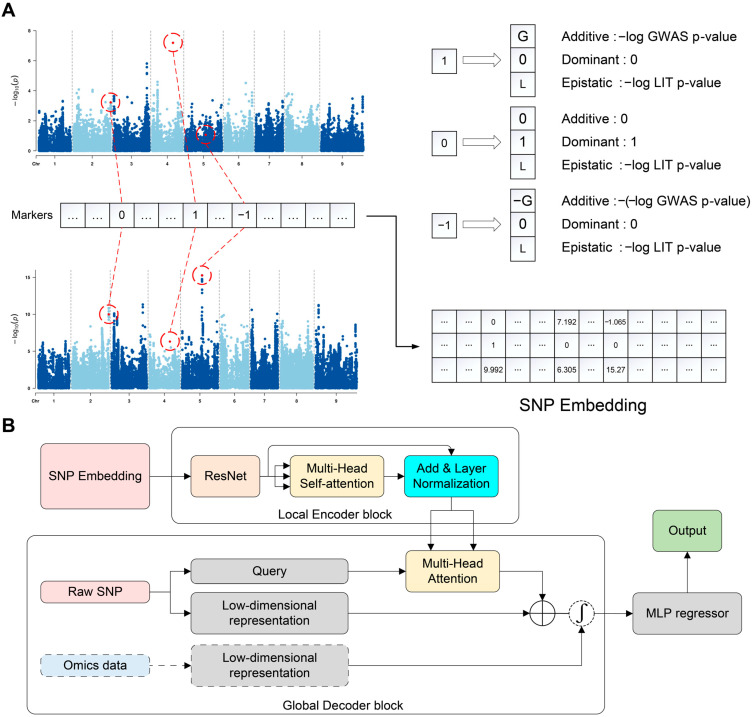
- Phần nhúng SNP tích hợp dữ liệu SNP với kết quả của nghiên cứu liên kết toàn bộ bộ gen (GWAS – Genome-Wide Association Study) và kiểm tra tương tác tiềm ẩn (LIT – Latent Interaction Testing) như kiến thức sinh học tiên nghiệm. Phương pháp nhúng này được thiết kế để mã hóa mỗi SNP chứa thông tin về hiệu ứng cộng gộp (additive), hiệu ứng trội (dominant) và hiệu ứng tương tác (epistatic).
- Bộ mã hóa cục bộ sử dụng khối ResNet để giảm chiều dữ liệu và mã hóa thông tin cục bộ.
- Bộ giải mã toàn cục sử dụng cơ chế chú ý đa đầu (multi-head attention mechanism) để trích xuất các đặc trưng toàn cục, đồng thời cung cấp một mô-đun cho phép kết hợp các dữ liệu omics khác (ví dụ: dữ liệu chuyển hóa).
- MLP được sử dụng làm bộ hồi quy để đưa ra kết quả dự đoán cuối cùng.
Kết quả và Đánh giá
- Thông qua các thử nghiệm trên bốn tập dữ liệu khác nhau (millet827, spruce1722, soy5014, maize5820), iADEP cho thấy hiệu quả vượt trội so với các phương pháp hiện có trong dự đoán kiểu gen thành kiểu hình.
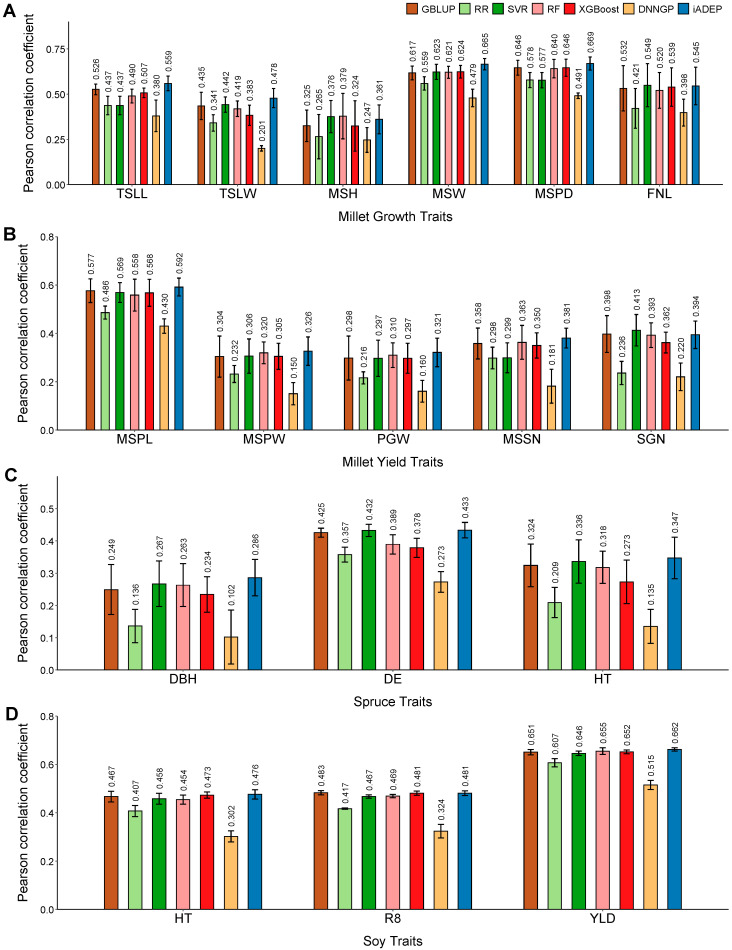
- Các thí nghiệm cắt bỏ (ablation experiments) đã xác nhận hiệu quả của phương pháp nhúng SNP. Các phương pháp mã hóa đơn giản hơn (one-hot encoding, non-encoding) thường dẫn đến hiệu quả dự đoán thấp hơn.
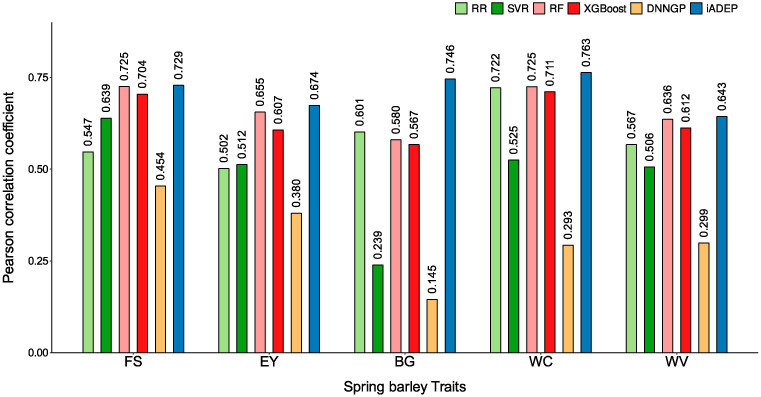
- iADEP có khả năng kết hợp dữ liệu omics khác. Khi tích hợp dữ liệu chuyển hóa trên tập dữ liệu springbarley2463, iADEP đạt hiệu quả dự đoán cao nhất so với các phương pháp khác (ngoại trừ GBLUP không tương thích).
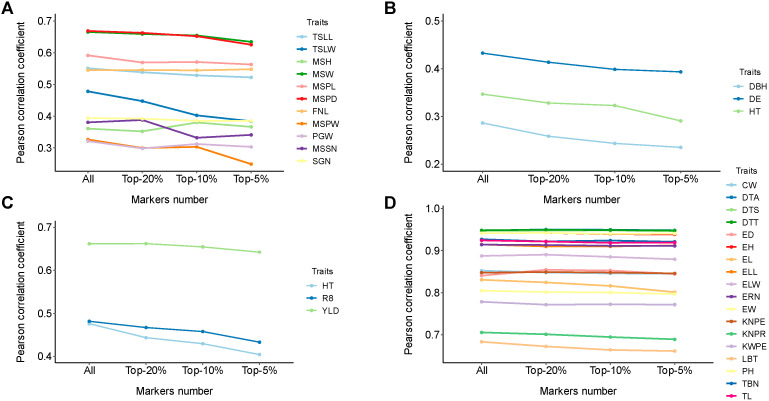
- Việc lựa chọn đặc trưng (feature selection) bằng cách chỉ sử dụng các SNP liên quan đến GWAS hoặc LIT thường không cải thiện hiệu quả dự đoán của iADEP. Sử dụng toàn bộ tập hợp SNP thường mang lại kết quả tối ưu cho iADEP trên hầu hết các tính trạng.
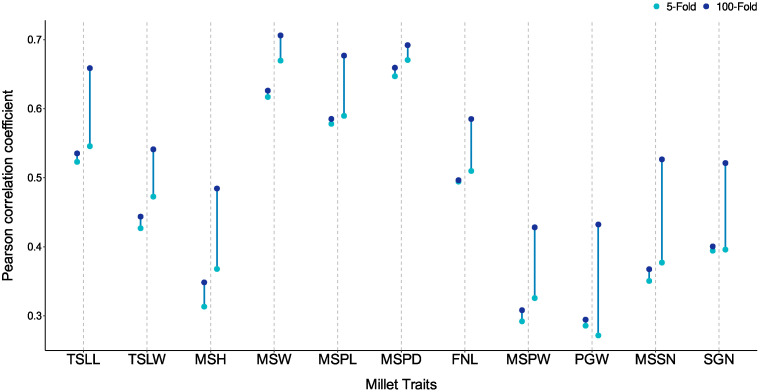
- Nghiên cứu sự khác biệt giữa học chuyển dịch (transductive learning – ví dụ GBLUP) và học quy nạp (inductive learning – ví dụ iADEP) cho thấy iADEP rất nhạy cảm với kích thước của tập huấn luyện. Hiệu quả của iADEP cải thiện đáng kể khi tăng kích thước tập huấn luyện (ví dụ: từ 5-fold sang 100-fold cross-validation), trong khi GBLUP ít nhạy cảm hơn. Điều này cho thấy iADEP sẽ hưởng lợi từ kỷ nguyên dữ liệu lớn.
Thảo luận và Hạn chế
- Vai trò tiềm năng của nhúng SNP là phân biệt các SNP dựa trên kiến thức sinh học (GWAS, LIT) và đóng vai trò như một dạng huấn luyện sơ bộ (pre-training).
- Khả năng mở rộng của iADEP cho phép kết hợp không chỉ dữ liệu omics mà tiềm năng cả dữ liệu môi trường trong tương lai.
- Mô hình học quy nạp của iADEP cung cấp nền tảng cho sự phát triển trong tương lai, đặc biệt là khả năng giải thích (interpretability).
- Tuy nhiên, iADEP có một số hạn chế: quá trình xây dựng mô hình kém linh hoạt hơn (cần điều chỉnh siêu tham số thủ công), yêu cầu kích thước tập huấn luyện lớn hơn để đạt hiệu quả cao, yêu cầu phần cứng cao hơn (GPU cho dữ liệu lớn), và tốn nhiều thời gian huấn luyện hơn so với GBLUP.
Kết luận
- iADEP là một mô hình học sâu mới đầy hứa hẹn cho dự đoán bộ gen thực vật, tích hợp hiệu quả kiến thức sinh học tiên nghiệm và cho phép kết hợp dữ liệu omics khác.
- Kết quả cho thấy hiệu suất dự đoán tốt của iADEP so với các phương pháp hiện có.
- Sự nhạy cảm của iADEP với kích thước dữ liệu huấn luyện chỉ ra rằng phương pháp dựa trên học sâu như iADEP sẽ được tăng cường sức mạnh bởi các bộ dữ liệu lớn trong tương lai.
- Những hiểu biết từ nghiên cứu iADEP góp phần vào việc ứng dụng rộng rãi học sâu trong chọn giống cây trồng, giúp tăng hiệu quả sản xuất và năng suất nông nghiệp, cũng như khả năng xác định các giống cây trồng thích nghi tốt hơn với môi trường.
- Sự phát triển của khả năng giải thích trong học sâu có thể tiết lộ thêm các cơ chế liên quan đến di truyền trong tương lai.
Tài liệu tham khảo: https://pmc.ncbi.nlm.nih.gov/articles/PMC12027452/#sec2-genes-16-00411
LOBI Vietnam là công ty tiên phong trong lĩnh vực Đọc trình tự gen thế hệ mới NGS (Next Generation Sequencing) và Phân tích Tin sinh học. Liên hệ hotline/Zalo 092.510.8899 để biết thêm chi tiết.




