Dự án lắp ráp hệ gen động vật có xương sống
Việc lắp ráp các hệ gen tham chiếu cung cấp một bản đồ trình tự của một loài sinh vật và “bối cảnh không gian” của nó – nghĩa là vị trí của một đoạn DNA cụ thể được tìm thấy dọc theo các nhiễm sắc thể. Trong quá khứ, việc xây dựng được các hệ gen tham chiếu rất tốn kém và đòi hỏi nhiều công sức, vì thế chúng chỉ được xây dựng cho một nhóm sinh vật quan trọng, trong đó có con người, đương nhiên vẫn còn các khoảng trống và các vùng bị lỗi sau lắp ráp chưa xử lý được. Các bộ gen được tạo ra nhờ công nghệ giải trình tự thứ 2 (short-reads) có thể được lắp ráp cho nhiều loài sinh vật, nhưng kết quả lắp ráp thấp do bị phân mảnh nhiều dẫn đến kết quả chú giải sai lệch ở nhiều vùng.
Tuy nhiên, để có 1 hiểu biết toàn diện về quá trình tiến hóa và trả lời được các câu hỏi cơ bản của sinh học thì việc tạo ra các hệ gen tham chiếu có chất lượng cao cho tất cả các loài sinh vật là cần thiết. Nhờ các tiến bộ công nghệ, tăng khả năng tính toán và giảm chi phí giải trình tự thì vào năm 2017, dự án VGP được khởi động. Dự án này ra đời nhằm theo đuổi một tham vọng lớn lao là tạo ra hệ gen tham chiếu cho từng loài động vật có xương sống còn tồn tại trên Trái Đất.
Dự án được chia làm 4 giai đoạn. Giai đoạn 1 tập trung thử nghiệm và cải thiện kết quả giải trình tự và lắp ráp hệ gene, đồng thời tiến hành lắp ráp 260 hệ gene đầu tiên kết hợp với các báo cáo ban đầu về sự tiến hóa hệ gene ở động vật có xương sống. Giai đoạn thứ 2 nhắm tới lắp ráp 1159 họ gene động vật, giai đoạn thứ 3 là 10000 chi. Cuối cùng, tất cả các loài đều được lắp ráp thành công.
Theo thống kê hiện nay, có khoảng 3 hệ gene lắp ráp được tạo ra mỗi tuần và sẽ tăng lên 125 hệ gen/tuần để đạt được mục tiêu này trong vòng 10 năm.
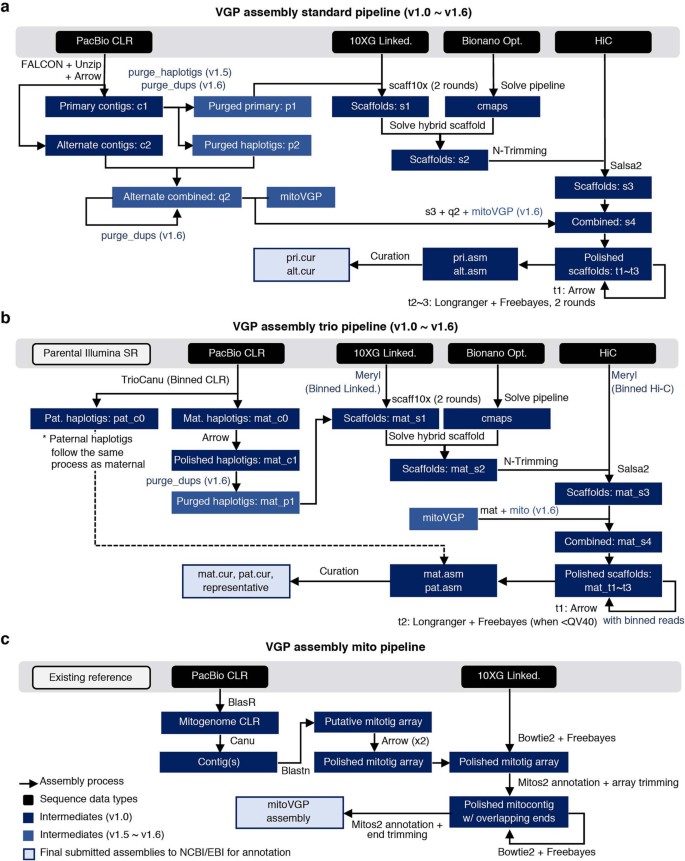
Tham khảo: https://www.nature.com/articles/s41586-021-03451-0
LOBI Vietnam là công ty tiên phong trong lĩnh vực Đọc trình tự gen thế hệ mới NGS (Next Generation Sequencing) và Phân tích Tin sinh học. Liên hệ hotline/Zalo 092.510.8899 để biết thêm chi tiết.


