MỤC LỤC BÀI VIẾT
Tổng quan và Đặt vấn đề
- Dữ liệu scRNA-seq cung cấp mô tả toàn diện về hồ sơ biểu hiện gen của mỗi tế bào đơn lẻ, giúp hiểu rõ hơn về sự phát triển và chức năng của các sinh vật sống. Mặc dù công nghệ giải trình tự đã tiến bộ, chi phí và khó khăn trong việc thu thập dữ liệu vẫn cao. Mẫu sinh học đôi khi khó thu được, và một số loại tế bào có thể quá hiếm để phân tích. Việc thu thập đủ dữ liệu scRNA-seq chất lượng cao vẫn là một thách thức lớn.
- Để giải quyết vấn đề này, một số nhà nghiên cứu đã cố gắng tạo dữ liệu biểu hiện gen in silico (trên máy tính). Có hai loại phương pháp chính: mô hình thống kê và mô hình tạo sinh sâu (deep generative models). Các mô hình thống kê (như ZINB, SPARSim, SCRIP, scDesign3) thường đơn giản hóa các mẫu phức tạp trong dữ liệu thực và khó tạo dữ liệu dưới các điều kiện không được thiết kế ban đầu.
- Sự phát triển gần đây của mô hình tạo sinh sâu mang đến cơ hội lớn cho việc tạo dữ liệu transcriptomic in silico. Các mô hình hiện tại chủ yếu dựa trên autoencoder biến phân (VAE) và mạng đối nghịch tạo sinh (GAN). Các mô hình VAE-based (như scVI) chủ yếu tập trung vào các nhiệm vụ phân tích hạ nguồn (ví dụ: sửa lỗi batch, phân cụm) hơn là tạo hồ sơ biểu hiện gen. Các mô hình GAN-based (như scGAN) có thể tạo tế bào mới, nhưng chỉ từ phân phối đã biết, không đáp ứng được nhu cầu bổ sung dữ liệu chưa đo lường. Hơn nữa, GAN yêu cầu thiết kế và tinh chỉnh phức tạp, gây khó khăn cho việc ứng dụng trên các tập dữ liệu mới và tạo dữ liệu dưới các điều kiện nhất định.
- Gần đây, mô hình khuếch tán tiềm ẩn (latent diffusion model – LDM) đã cho thấy hiệu suất xuất sắc trong nhiều lĩnh vực. So với GAN, LDM có quá trình huấn luyện ổn định và dễ dàng tạo mẫu dựa trên các điều kiện phức tạp. Tuy nhiên, ít nghiên cứu ứng dụng LDM trong lĩnh vực đơn bào. Một thách thức là LDM cần một mô hình autoencoder được tiền huấn luyện để liên kết dữ liệu trong không gian tiềm ẩn và không gian gốc. Sự xuất hiện gần đây của các mô hình nền tảng (foundation models) đã giải quyết thách thức này, vì chúng có thể được sử dụng làm mô hình autoencoder trong LDM.
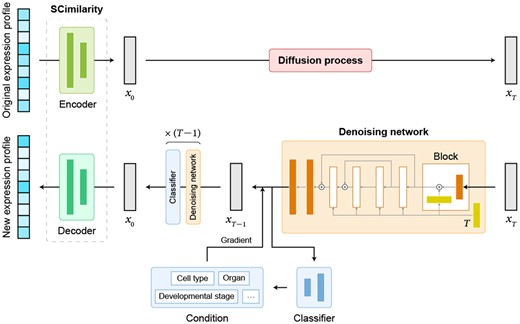
- Để giải quyết các vấn đề trên, các tác giả đề xuất scDiffusion, một mô hình tạo dữ liệu scRNA-seq in silico kết hợp LDM với mô hình nền tảng để tạo dữ liệu biểu hiện gen đơn bào với các điều kiện cho trước. scDiffusion có ba phần: một autoencoder, một mạng khử nhiễu (denoising network) và một bộ điều khiển điều kiện (condition controller). Mô hình nền tảng được tiền huấn luyện SCimilarity được sử dụng làm autoencoder để chuẩn hóa phân phối thô và giảm chiều dữ liệu scRNA-seq. Mạng khử nhiễu được thiết kế lại dựa trên mạng perceptron đa lớp (MLP) có kết nối nhảy (skip-connected). Bộ điều khiển điều kiện là một bộ phân loại loại tế bào (cell type classifier), cho phép scDiffusion tạo dữ liệu đặc trưng cho một loại tế bào hoặc cơ quan cụ thể theo yêu cầu.
Mô hình scDiffusion
- Mô hình scDiffusion gồm ba phần: mô hình nền tảng SCimilarity đã được tiền huấn luyện làm autoencoder, mạng khử nhiễu và bộ phân loại điều kiện
Tinh chỉnh mô hình nền tảng được tiền huấn luyện làm autoencoder
Một điều kiện tiên quyết cho LDM là có autoencoder phù hợp để nối dữ liệu trong không gian tiềm ẩn và không gian gốc. Các tác giả đã sử dụng mô hình SCimilarity được tiền huấn luyện làm autoencoder. SCimilarity là mạng kiểu mã hóa-giải mã (encoder-decoder) được huấn luyện trên tập dữ liệu lớn gồm 22.7 triệu tế bào. Mô hình SCimilarity được tinh chỉnh trên dữ liệu của họ. Đầu vào của bộ mã hóa là hồ sơ biểu hiện gen đã được chuẩn hóa và logarithm, đầu ra là embedding không gian tiềm ẩn 128D. Bộ giải mã sau đó nhận embedding và tạo ra hồ sơ biểu hiện tương ứng. Việc sử dụng trọng số tiền huấn luyện giúp tiếp cận autoencoder mong muốn nhanh hơn và tốt hơn so với huấn luyện từ đầu. Autoencoder giúp chuyển đổi phân phối dữ liệu biểu hiện gen sang phân phối giống Gaussian, phù hợp với quá trình khuếch tán.
Huấn luyện mạng khử nhiễu
Sau khi có embedding từ bộ mã hóa, quá trình khuếch tán được áp dụng cho từng embedding để tạo dữ liệu nhiễu. Mạng khử nhiễu được huấn luyện trên các embedding nhiễu này để học quá trình khuếch tán ngược. Vì hồ sơ biểu hiện gen là vector dài, thưa thớt và không có thứ tự, các mạng khử nhiễu cổ điển như CNN không phù hợp. Các tác giả đã phát triển kiến trúc mới với các lớp kết nối đầy đủ (fully connected layers) và cấu trúc kết nối nhảy (skip-connected structure). Cấu trúc kết nối nhảy giúp duy trì đặc điểm của các đặc trưng ở các cấp độ khác nhau. Quá trình khuếch tán xuôi thêm nhiễu vào embedding gốc (x0) qua T bước để tạo embedding nhiễu (xT). Mục tiêu huấn luyện là học quá trình khuếch tán ngược để dự đoán xi-1 từ xi. Mạng xương sống (backbone network) dự đoán nhiễu được thêm vào. Trong quá trình suy luận (inference), mô hình khuếch tán nhận nhiễu Gaussian làm đầu vào ban đầu và khử nhiễu lặp đi lặp lại qua T bước để thu được embedding không gian tiềm ẩn mới (x0), sau đó được đưa vào bộ giải mã để có dữ liệu biểu hiện gen cuối cùng.
Tạo có điều kiện và chiến lược Gradient Interpolation
Các tác giả đã đề xuất chiến lược Gradient Interpolation để tạo điều hướng điều kiện tế bào liên tục. Một bộ phân loại nhận hai điều kiện khác nhau (ví dụ: trạng thái ban đầu và cuối của biệt hóa tế bào) và tạo ra hai gradient cùng lúc. Các gradient này sau đó được tích hợp để điều hướng quá trình khuếch tán đến trạng thái trung gian chưa từng thấy. Bằng cách điều chỉnh các hệ số γ1 và γ2, scDiffusion có thể quyết định tế bào được tạo ra sẽ gần với trạng thái tế bào nào hơn, từ đó tạo ra các tế bào với trạng thái liên tục. Với chiến lược này, trạng thái ban đầu của quá trình tạo sinh khuếch tán được thay đổi từ nhiễu Gaussian thuần túy sang embedding không gian tiềm ẩn của các tế bào điều kiện ban đầu, theo một quá trình thêm nhiễu. Điều chỉnh này giữ lại các đặc điểm chung của các tế bào ban đầu, cho phép mô hình tạo ra một loạt các trạng thái tế bào mới cho mỗi trạng thái ban đầu được cung cấp, từ đó tạo thành một quỹ đạo trạng thái tế bào liên tục.
Kết quả và Đánh giá
Tạo dữ liệu scRNA-seq thực tế
scDiffusion được áp dụng trên các tập dữ liệu Tabular Muris, Human Lung PF và PBMC68k để tạo tế bào mới mà không có điều hướng của bộ phân loại. So sánh với scGAN và scDesign3. Các chỉ số (SCC, MMD, LISI, AUC của Random Forest) cho thấy scDiffusion có thể tạo dữ liệu scRNA-seq thực tế tương đương với các phương pháp hiện đại và vượt trội hơn SPARSim và SCRIP. Việc sử dụng mô hình nền tảng được tiền huấn luyện làm autoencoder giúp đạt chất lượng tạo dữ liệu cao hơn với thời gian huấn luyện ngắn hơn

Tạo có điều kiện các loại tế bào cụ thể
Một bộ phân loại loại tế bào được huấn luyện trên tập dữ liệu Tabular Muris để điều hướng tạo dữ liệu có điều kiện. Các tế bào được tạo ra theo điều kiện trực quan trùng lặp với các tế bào thực trên biểu đồ UMAP. So sánh với cscGAN và scDesign3. Phân loại CellTypist cho thấy tế bào được tạo ra bởi scDiffusion có độ chính xác phân loại gần với tế bào thực, trong khi tế bào tạo ra bởi cscGAN không thể phân biệt được. Mô hình KNN cho thấy không thể phân biệt giữa tế bào được tạo ra bởi scDiffusion của một loại cụ thể và tế bào thực cùng loại (AUC gần 0.5), trong khi có thể dễ dàng phân biệt tế bào tạo ra bởi GAN và scDesign3. Biểu hiện của các gen đánh dấu chính trong dữ liệu Tabular Muris được tạo ra bởi scDiffusion cũng gần với dữ liệu thực. Kết quả tương tự được quan sát với tập dữ liệu PBMC68k. Các loại tế bào hiếm (như Thymus cell trong Tabular Muris và CD34+ cell trong PBMC68k) cũng có thể được tạo ra tốt.

Tạo dữ liệu tế bào out-of-distribution với nhiều điều kiện
Các tác giả đã thử tạo tế bào với nhiều điều kiện dựa trên tập dữ liệu Tabular Muris, sử dụng hai bộ phân loại riêng biệt cho loại cơ quan và loại tế bào. Bằng cách kết hợp các điều kiện đã biết (ví dụ: T cell tuyến vú, T cell lách, B cell lách), scDiffusion có thể tạo ra loại tế bào có tổ hợp điều kiện mới (B cell tuyến vú) chưa từng thấy trong dữ liệu huấn luyện (out-of-distribution). Phân loại CellTypist cho thấy tế bào B tuyến vú được tạo ra bởi scDiffusion có độ chính xác phân loại cao, tương đương với tế bào thực. Biểu hiện của các gen đánh dấu (CD74, CD79A) trong tế bào được tạo ra tương tự như tế bào thực. Thí nghiệm tương tự với tập dữ liệu Tabular Sapiens cũng cho kết quả khả quan trong việc tạo ra tế bào memory B lách và tế bào macrophage lách, vốn đã bị loại bỏ khỏi dữ liệu huấn luyện. Biểu hiện của các gen đánh dấu của các loại tế bào này cũng tương tự như tế bào thực. Kết quả cho thấy scDiffusion có thể tạo ra các tế bào out-of-distribution thực tế bằng cách học các mẫu biểu hiện của các tế bào đã biết.

Tạo trạng thái tế bào trung gian trong quá trình tái lập trình tế bào
Chiến lược Gradient Interpolation được sử dụng để tạo các trạng thái tế bào trung gian trong quá trình tái lập trình tế bào (MEF sang iPSC) trong tập dữ liệu Waddington-OT. scDiffusion được huấn luyện trên dữ liệu từ ngày 0 đến ngày 8 (loại bỏ ngày 3.5 và 4). Bằng cách sử dụng Gradient Interpolation với hai điều kiện đầu vào (ngày 3 và ngày 4.5), scDiffusion có thể tạo ra một loạt các trạng thái tế bào giữa ngày 3 và ngày 4.5 trên quỹ đạo phát triển. So sánh với nội suy tuyến tính (linear interpolation), scDiffusion cho thấy hiệu suất tốt hơn ở các chỉ số MMD và LISI, ngay cả khi không được huấn luyện với thông tin về các nhóm xử lý khác nhau trong dữ liệu. Các trạng thái trung gian được tạo ra bởi scDiffusion gần với các tế bào thực bị loại khỏi dữ liệu huấn luyện hơn so với nội suy tuyến tính. scDiffusion cũng bắt được các thay đổi phi tuyến tính của các gen đánh dấu chính tốt hơn, có khả năng hỗ trợ khám phá sinh học ở các trạng thái trung gian.

Thảo luận và Hạn chế
- scDiffusion là một mạng nơ-ron tạo sinh sâu dựa trên LDM và mô hình nền tảng. Nó có thể được huấn luyện ổn định trên tập dữ liệu lớn và tạo ra dữ liệu thực tế. scDiffusion có thể tạo hồ sơ biểu hiện gen dưới bất kỳ điều kiện nào ở cấp độ đơn bào, kể cả đối với các loại tế bào hiếm. Sử dụng khả năng tạo đa điều kiện và chiến lược Gradient Interpolation, scDiffusion có khả năng độc đáo trong việc tạo dữ liệu out-of-distribution cũng như các trạng thái trung gian giữa hai trạng thái tế bào đã biết. Chức năng này được cho là độc đáo đối với scDiffusion
- Cần thêm nỗ lực để thiết kế các chỉ số đánh giá tốt hơn để phù hợp với sự phức tạp ngày càng tăng của dữ liệu được tạo ra. Một chỉ số đánh giá toàn diện và áp dụng phổ quát là rất cần thiết. Các chỉ số hiện tại dựa trên dữ liệu đã biết và có thể không đủ để đánh giá dữ liệu chưa từng thấy. Việc xác định ngưỡng độ tin cậy cho dữ liệu mới vẫn là thách thức.
Kết luận
scDiffusion có thể được áp dụng trong nhiều ứng dụng hạ nguồn khác. Một ứng dụng tự nhiên là tạo dữ liệu đa omics. Về lý thuyết, scDiffusion có thể tạo ra bất kỳ loại dữ liệu đơn bào nào. Ngoài ra, scDiffusion cũng có thể được sử dụng để cải thiện chất lượng dữ liệu đơn bào, ví dụ như khử nhiễu cho dữ liệu bị nhiễm bẩn. Trong tương lai, có thể thay thế bộ phân loại bằng các công cụ mạnh mẽ hơn như CLIP để kiểm soát quá trình tạo sinh bằng các điều kiện phức tạp hơn, cho phép các nhiệm vụ phức tạp hơn như can thiệp tế bào in silico, hỗ trợ quan trọng cho việc lựa chọn thuốc và kiểm soát quá trình chuyển đổi trạng thái tế bào
Tài liệu tham khảo:
https://academic.oup.com/bioinformatics/article/40/9/btae518/7738782
LOBI Vietnam là công ty tiên phong trong lĩnh vực Đọc trình tự gen thế hệ mới NGS (Next Generation Sequencing) và Phân tích Tin sinh học. Liên hệ hotline/Zalo 092.510.8899 để biết thêm chi tiết.




