- Giới thiệu và Vấn đề cần giải quyết
Mạng lưới điều hòa gen (GRN) mô tả các mối quan hệ điều hòa phức tạp giữa các gen và là một trong những công cụ chính giúp các nhà nghiên cứu phân tích và hiểu các quá trình sinh học ở cấp độ phân tử1. Suy luận các mối quan hệ điều hòa này một cách chính xác từ dữ liệu biểu hiện gen đã trở thành trọng tâm nghiên cứu trong sinh học tính toán và tin sinh học, có ý nghĩa quan trọng trong y sinh học và khám phá các quá trình sinh học tiềm năng. Ban đầu, các nhà nghiên cứu sử dụng các phương pháp dựa trên thống kê truyền thống để suy luận GRN. Tuy nhiên, các phương pháp này chỉ xem xét các mẫu thống kê tồn tại trong dữ liệu biểu hiện gen và bỏ qua mối quan hệ nguyên nhân kết quả giữa dữ liệu biểu hiện gen. Điều này dẫn đến kết quả suy luận có độ chính xác thấp và thiếu ý nghĩa sinh học. Do đó, các nhà nghiên cứu bắt đầu tập trung vào việc phân tích các mối quan hệ điều hòa nguyên nhân – kết quả giữa các gen. Các phương pháp suy luận mạng nguyên nhân – kết quả truyền thống đã được đề xuất, chẳng hạn như mô hình vi phân phi tuyến dựa trên dữ liệu chuỗi thời gian…, mạng Bayesian…, mạng Bayesian động…, suy luận cạnh nguyên nhân – kết quả bằng phân tích ảnh hưởng…, hay sử dụng Entropy Truyền (Transfer Entropy – TE) để suy luận khung mạng và lọc cạnh…. Các phương pháp này đã cải thiện độ chính xác và tạo ra kết quả có ý nghĩa sinh học. Tuy nhiên, các phương pháp suy luận mạng nguyên nhân – kết quả truyền thống gặp phải hai vấn đề chính: thời gian và tài nguyên tính toán quá mức… (đặc biệt khi số điểm thời gian tăng lên đối với mạng Bayesian động…) và thiếu các ràng buộc hiệu quả trong quá trình suy luận, có thể dẫn đến việc tạo ra kết quả sai lệch.
- Ứng dụng Học sâu và Hạn chế của GCN truyền thống
Với sự tiến bộ của học sâu, các nhà nghiên cứu đã bắt đầu sử dụng các phương pháp học sâu để suy luận GRN hiệu quả hơn…. So với các phương pháp suy luận mạng nguyên nhân – kết quả truyền thống, các phương pháp học sâu có thể học các mối quan hệ điều hòa phức tạp hơn từ dữ liệu biểu hiện với độ chính xác cao hơn.
Mạng Nơ-ron Đồ thị (GNN) là mô hình mạng có khả năng xử lý dữ liệu đồ thị, cho phép biểu diễn và suy luận cấu trúc đồ thị một cách hiệu quả…. Do đó, việc sử dụng mạng nơ-ron đồ thị để khai thác cấu trúc liên kết điều hòa phức tạp hơn dựa trên các mối quan hệ điều hòa gen đã biết dần trở thành một công cụ mới để suy luận GRN…. Một số phương pháp đã sử dụng GNN cho dự đoán liên kết… hoặc mạng chú ý đồ thị (Graph Attention Network)…. Tuy nhiên, cơ chế chú ý dựa trên đồ thị có thể gây ra chi phí tính toán và bộ nhớ lớn….
Mạng Tích chập Đồ thị (GCN) là một phương pháp dựa trên đồ thị được xây dựng trên GNN…. Thông qua các phép toán tích chập và tổng hợp theo cấp bậc, GCN ổn định và chính xác hơn GNN trong việc tạo ra tổng hợp lân cận…. GCN đã được sử dụng rộng rãi trong các mạng lưới sinh học. Một số phương pháp đã kết hợp GCN với Autoencoder biến phân đồ thị (GVAE)… hoặc sử dụng bộ mã hóa tương tác dựa trên GCN….
Tuy nhiên, một hạn chế cố hữu của GCN là thông tin lân cận dễ bị mất trong quá trình tổng hợp nhiều lớp…. Việc cập nhật liên tục thông tin lân cận dẫn đến mất mát một số thông tin gốc. Do đó, biểu diễn đặc trưng nút cuối cùng khó có thể nắm bắt đầy đủ toàn bộ đồ thị, dẫn đến độ chính xác không đáng tin cậy trong các nhiệm vụ tiếp theo….
Vì vậy, chìa khóa để suy luận GRN bằng mạng nơ-ron đồ thị là đảm bảo các mối quan hệ nguyên nhân – kết quả được nhúng trong các cặp điều hòa đã biết và dữ liệu biểu hiện không bị bỏ qua trong quá trình tổng hợp các nút lân cận.
- Phương pháp đề xuất: GCN dựa trên Tái cấu trúc Đặc trưng Nguyên nhân – Kết quả(CRGCN)
Để giải quyết vấn đề mất mát thông tin và tăng cường mối quan hệ nguyên nhân – kết quả, bài viết đề xuất một phương pháp mới để suy luận GRN sử dụng GCN được hướng dẫn bởi thông tin nguyên nhân – kết quả….
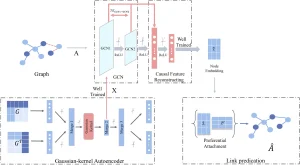
Phương pháp này sử dụng Transfer Entropy (TE) và lớp tái cấu trúc để đạt được tái cấu trúc đặc trưng nguyên nhân – kết quả. Mục tiêu là giảm thiểu vấn đề mất mát thông tin do nhiều vòng tổng hợp lân cận trong GCN, tạo ra biểu diễn đặc trưng nút nguyên nhân – kết quả và toàn diện…. Điều này giúp tăng cường tính nguyên nhân – kết quả và sự phụ thuộc giữa các lân cận ở mỗi lớp….
Mô hình đề xuất, gọi là CRGCN, bao gồm ba thành phần chính:
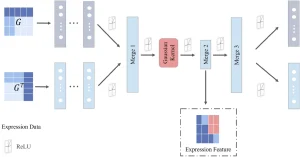
Mô-đun Gaussian-kernel Autoencoder (gAE)…:
Mục đích: Trích xuất các đặc trưng biểu hiện gen có khả năng phân tách đáng kể (significantly separable features) từ dữ liệu biểu hiện gen thô…. Điều này nhằm cải thiện hiệu quả tính toán và độ chính xác của tái cấu trúc nguyên nhân – kết quả sau này…. Việc trích xuất các đặc trưng hiệu quả từ dữ liệu biểu hiện là rất quan trọng để suy luận GRN chính xác. gAE có khả năng khai thác các đặc trưng sâu hơn, phức tạp hơn so với các phương pháp khác như GKF, SVD, NMF.
Cách hoạt động: Tích hợp nhân Gaussian vào cấu trúc Autoencoder…. Dữ liệu biểu hiện gen và dạng chuyển vị của nó được sử dụng làm đầu vào để trích xuất các đặc trưng điều hòa được nhúng trong cả hàng và cột. Các đặc trưng được tăng cường bằng nhân Gaussian để trở nên khác biệt/phân tách hơn…. Sử dụng các lớp trộn (merge layers) dựa trên MLP để mã hóa và kết hợp sâu hơn các đặc trưng này. Mô-đun nhân Gaussian giúp nắm bắt các đặc trưng khác biệt. Hàm lỗi kết hợp Sai số Bình phương Trung bình (MSE) và KL divergence để đảm bảo đặc trưng trích xuất phản ánh chính xác các đặc trưng sâu. Tham số σ của nhân Gaussian ảnh hưởng đến khả năng phân tách đặc trưng…; thử nghiệm cho thấy σ = 1 mang lại kết quả tốt nhất….
Mô-đun GCN dựa trên tái cấu trúc đặc trưng nguyên nhân – kết quả…:
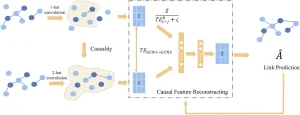
- Quá trình: Nhận đặc trưng biểu hiện (X) từ gAE và ma trận kề (A) từ các cặp điều hòa đã biết làm đầu vào.
- Lớp tích chập đồ thị: Thực hiện tổng hợp lân cận để tạo ra biểu diễn đặc trưng nút mới…. Nhiều lớp giúp thu thập thông tin lân cận ở tất cả các bậc….
- Tái cấu trúc đặc trưng nguyên nhân – kết quả: Sử dụng Transfer Entropy (TE) để định lượng mức độ mất mát thông tin nguyên nhân – kết quả trong quá trình tổng hợp lân cận…. Giá trị TE cho thấy sức mạnh của mối quan hệ nguyên nhân – kết quả. Thông tin lân cận có mức độ chấp nhận thấp hơn (TE thấp hơn) sẽ được trọng số hóa nhiều hơn…. Thông tin lân cận đã được trọng số hóa ở mỗi bậc được kết hợp trong mô-đun tái cấu trúc (sử dụng MLP) để thu được biểu diễn đặc trưng nút nguyên nhân – kết quả toàn diện và chính xác hơn…. KL divergence được sử dụng để giảm thiểu sai khác tái cấu trúc…. Đặc trưng tái cấu trúc Ẑ là đầu ra….
Mô-đun dự đoán liên kết (Link prediction)…:
- Mục đích: Từ các đặc trưng nút đã được tái cấu trúc nguyên nhân – kết quả, suy luận các mối quan hệ điều hòa gen (dự đoán các cạnh trong mạng)….
- Phương pháp: Sử dụng phương pháp Ưu tiên Gắn kết (Preferential Attachment – PA) để dự đoán điểm tương đồng giữa các đặc trưng nút. PA hiệu quả về mặt tính toán, hoạt động tốt trên các mạng lưới liên kết dày đặc và đặc biệt phù hợp với GRN.
- Đầu ra: Dựa trên điểm tương đồng, mô hình tạo ra ma trận kề của mạng dự đoán (GRN đã được suy luận)….
- Thực nghiệm và Kết quả
Phương pháp được kiểm tra trên các bộ dữ liệu DREAM5 (E.coli, S.cerevisiae)… và mDC (Mouse dendritic cell)…. Bộ dữ liệu S.cerevisiae có sự mất cân bằng lớp (ít cạnh đúng hơn cạnh sai). Chi tiết bộ dữ liệu được cung cấp trong Bảng 134….
Các chỉ số đánh giá chính là AUROC (Area Under the Receiver Operating Characteristic Curve) và AUPRC (Area Under the Precision–Recall Curve). AUPRC được coi trọng hơn trong suy luận GRN, đặc biệt đối với mạng mất cân bằng lớp.
Các thử nghiệm được thiết lập để xác minh hiệu quả của từng thành phần và hiệu quả tổng thể:
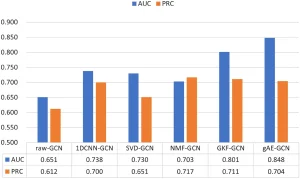
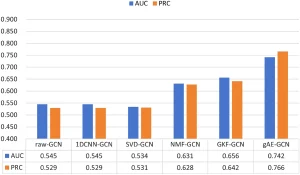
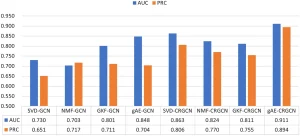
Thử nghiệm 1: Xác thực hiệu quả của các phương pháp trích xuất đặc trưng…. So sánh gAE với các phương pháp khác (dữ liệu gốc, GKF, SVD, NMF, 1DCNN) khi sử dụng GCN 2 lớp cho dự đoán liên kết…. Kết quả (Hình 5-7) cho thấy gAE đạt chỉ số AUROC và AUPRC cao nhất trên E.coli và mDC…, và AUROC cao nhất trên S.cerevisiae.
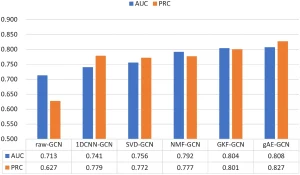


Điều này chứng tỏ gAE hiệu quả trong việc trích xuất các đặc trưng phân tách, sâu hơn và phức tạp hơn…. Thử nghiệm tham số σ của nhân Gaussian (Bảng 2, Hình 8-12) xác nhận σ = 1 là tối ưu cho việc trích xuất đặc trưng phân tách….




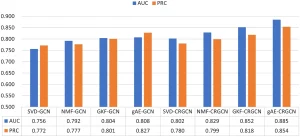
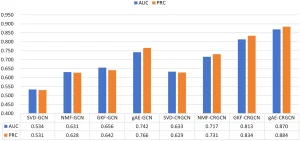
Thử nghiệm 2: Xác thực hiệu quả của tái cấu trúc đặc trưng nguyên nhân – kết quả. So sánh hiệu suất dự đoán liên kết khi sử dụng các phương pháp trích xuất đặc trưng (SVD, NMF, GKF, gAE) kết hợp với GCN thông thường so với CRGCN…. Kết quả (Hình 13-15) cho thấy việc sử dụng tái cấu trúc đặc trưng nguyên nhân – kết quả (CRGCN) cải thiện đáng kể cả AUROC và AUPRC trên tất cả các bộ dữ liệu và phương pháp trích xuất đặc trưng, đặc biệt là khi kết hợp với gAE. gAE-CRGCN cho thấy sự cải thiện đáng kể về AUROC và AUPRC so với gAE-GCN trên cả ba bộ dữ liệu (E.coli, S.cerevisiae, mDC). Điều này chứng tỏ tái cấu trúc đặc trưng nguyên nhân – kết quả giúp GCN thu được biểu diễn đặc trưng nút toàn diện hơn bằng cách tăng cường mối quan hệ nguyên nhân – kết quả giữa các nút lân cận ở mỗi lớp.
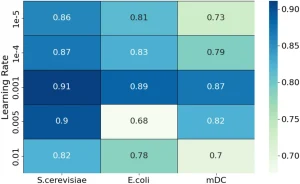
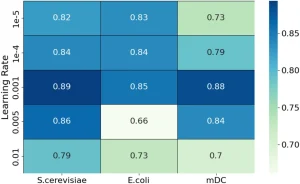
Thử nghiệm 3: Xác thực hiệu quả dự đoán liên kết tổng thể của gAE-CRGCN. Điều chỉnh tốc độ học (learning rate) cho thấy 0.001 là tối ưu (Hình 16, 17).
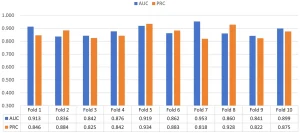
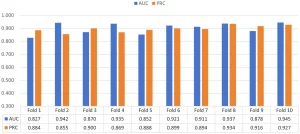
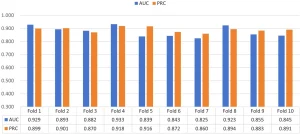
Thử nghiệm kiểm định chéo 10 lần (10-fold cross-validation) trên cả ba bộ dữ liệu (Hình 18-20) chứng minh hiệu suất và khả năng tổng quát hóa của gAE-CRGCN là ổn định….
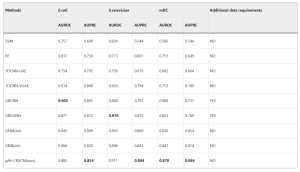
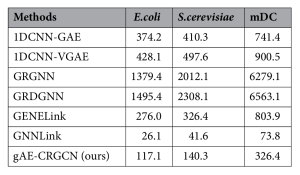
So sánh với các thuật toán hiện có: So sánh hiệu suất của gAE-CRGCN với nhiều phương pháp hiện có (SVM, RF, 1DCNN-GAE, 1DCNN-VGAE, GRGNN, GRDGNN, GENELink, GNNLink) (Bảng 3)….
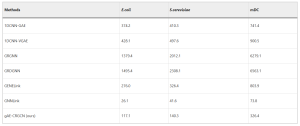
gAE-CRGCN đạt chỉ số AUPRC cao nhất trên cả ba bộ dữ liệu (E.coli, S.cerevisiae, mDC)…, cho thấy độ chính xác dự đoán vượt trội, đặc biệt hữu ích cho các mạng lưới mất cân bằng lớp. Mặc dù AUROC có thể hơi thấp hơn một số phương pháp cần dữ liệu bổ sung (GRGNN, GRDGNN), AUPRC lại cao hơn đáng kể, và AUPRC được coi trọng hơn trong suy luận GRN.
Thời gian tính toán: Phương pháp gAE-CRGCN có thời gian chạy thấp thứ hai trong số các phương pháp dựa trên mạng đồ thị được so sánh (Bảng 4)…, cho thấy đạt hiệu suất tốt hơn với chi phí tính toán thấp hơn.
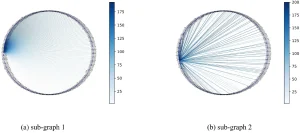
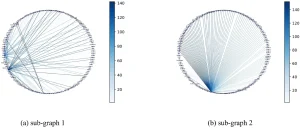
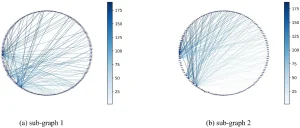
Kết quả thực nghiệm chứng minh gAE-CRGCN tăng cường tổng hợp nút ở mỗi lớp, tạo biểu diễn đặc trưng nút chi tiết và toàn diện hơn bằng cách kết hợp các đặc trưng đã được gAE kết hợp và tăng cường. Điều này dẫn đến độ tương đồng cao hơn trong dự đoán liên kết và cải thiện độ chính xác suy luận mạng. Các mạng con được suy luận cũng được trực quan hóa (Hình 21-23) để hiển thị chi tiết cấu trúc mạng….
- Kết luận và Hạn chế/Hướng phát triển
Bài viết kết luận rằng phương pháp GCN dựa trên tái cấu trúc đặc trưng nguyên nhân – kết quả cung cấp biểu diễn nút nguyên nhân – kết quả và toàn diện, giúp giảm thiểu sự mất mát thông tin trong quá trình tổng hợp lân cận. Việc kết hợp Gaussian-kernel Autoencoder giúp trích xuất các đặc trưng có khả năng phân tách đáng kể từ dữ liệu biểu hiện gen, cải thiện hiệu quả tính toán và độ tin cậy của tái cấu trúc đặc trưng nguyên nhân – kết quả, từ đó nâng cao độ chính xác của suy luận GRN. Các thử nghiệm trên bộ dữ liệu DREAM và mDC chứng minh phương pháp đề xuất đạt độ chính xác dự đoán vượt trội (đặc biệt là AUPRC cao nhất) và giảm thiểu thành công hạn chế của GCN trong việc bảo tồn thông tin lân cận, dẫn đến GRN được suy luận hợp lý, chính xác và đáng tin cậy hơn.
Tuy nhiên, mô hình vẫn bị ảnh hưởng bởi sự mất cân bằng lớp (class imbalance) và thiếu dữ liệu các cặp điều hòa đã biết. Hướng nghiên cứu trong tương lai bao gồm việc khai thác triệt để kiến thức tiên nghiệm về các mối quan hệ điều hòa gen để cải thiện hiệu suất trên các mạng lưới mất cân bằng lớp. Ngoài ra, việc khám phá và tích hợp dữ liệu đa omics được xem là một cách để mở rộng thông tin về dữ liệu sinh học và các mối quan hệ điều hòa.
LOBI Vietnam là công ty tiên phong trong lĩnh vực Đọc trình tự gen thế hệ mới NGS (Next Generation Sequencing) và Phân tích Tin sinh học. Liên hệ hotline/Zalo 092.510.8899 để biết thêm chi tiết.


