MỤC LỤC BÀI VIẾT
Tại sao lại cần nghiên cứu Metagenomics?
Metagenomics có thể được định nghĩa như là các kỹ thuật và phương pháp dùng để phân tích DNA tổng số của các quần xã vi sinh vật trong một môi trường cụ thể. Metagenomics có rất nhiều ứng dụng tiềm năng trong cả y học và vi sinh vật môi trường. Nghiên cứu Metagenomics được thực hiện nhiều nhất trong vi sinh môi trường là nghiên cứu về đa dạng của các quần xã vi sinh vật trong môi trường cụ thể thông qua phân tích các gene rRNA và đánh giá xem các quần xã vi sinh vật này thay đổi thế nào khi điều kiện lý hóa của môi trường biến đổi.
Metagenomics được ứng dụng nghiên cứu trong chăm sóc sức khỏe khi cơ thể con người có rất nhiều hệ vi sinh vật sinh sống, trên da, trong ruột, khoang miệng, khoang mũi, mắt, …. Các vi sinh vật này có ảnh hưởng lớn đến sức khỏe, hiệu quả sử dụng thuốc, khả năng hấp thu dinh dưỡng, … của bạn. Các nghiên cứu ước tính rằng
- số lượng vi sinh vật có trên cơ thể lớn gấp 10 lần số tế bào mà cơ thể bạn có,
- số lượng gene của vi sinh vật lớn gấp 100 lần số lượng gene mà bạn có
- và có hơn 1000 loài vi sinh vật khác nhau trên cơ thể bạn.

Giải trình tự thế hệ mới: Shotgun và Amplicon
| Shotgun Sequencing | Amplicon Sequencing |
|
|
Quy trình phân tích
Quy trình phân tích được chia làm 2 nhánh tương ứng với 2 hướng tiếp cận: Amplicon và Shotgun. Workflow của từng quy trình như hình bên dưới.

Tiền xử lý dữ liệu đọc trình tự
Có rất nhiều cách để lọc và cắt bỏ trình tự chưa tốt đối với dữ liệu NGS. Và bạn luôn phải thử đúng sai và chấp nhận đánh đổi giữa chất lượng và thông tin. Để đạt được chất lượng cao, bạn cần lọc bỏ nhiều trình tự hơn, đồng nghĩa là bạn cũng mất đi nhiều thông tin hơn.
Lưu ý rằng, loại bỏ các Chimera reads là cần thiết do trong quá trình PCR, các trình tự có thể gộp lại tạo ra trình tự lai.


Phân cụm OTU
Từ dữ liệu sau tinh sạch, nhiệm vụ của chuyên viên phân tích tin sinh học sau đó là xác định các loài và kiểm đếm sự xuất hiện của các loài. Do bản chất không đầy đủ của phân loại vi khuẩn và sự hiện diện của các lỗi trình tự trong các lần đọc NGS, một cách tiếp cận phổ biến là phân cụm các trình tự read có một mức độ tương đồng nào đó thành các chuỗi đại diện của loài giả được gọi là Đơn vị phân loại hoạt động (Operational Taxonomic Units – OTU). Các trình tự có mức độ tương đồng cao (ví dụ là > 97%) được nhóm lại với nhau và được đại diện bởi một chuỗi OTU duy nhất. Đầu ra chính của quy trình phân nhóm và kiểm đếm là một bảng OTU, liệt kê sự phong phú của các OTU trong các mẫu đang điều tra. Các phân tích thứ cấp bao gồm các ước tính về sự đa dạng alpha và beta trong bối cảnh siêu dữ liệu mẫu, ngoài các thử nghiệm thống kê về mức độ phong phú khác biệt.

Tìm kiếm tương đồng trong cơ sở dữ liệu marker, taxonomy và chức năng
Để xác định loài, các trình tự sẽ được tìm kiếm tương đồng trong các cơ sở dữ liệu tham chiếu cho Amplicon (green genes và silva) và shotgun (MetaPhlAn2) . Mức độ chính xác của kết quả phụ thuộc hoàn toàn vào mức độ đầy đủ của các CSDL. Thế nhưng, một điều chắc chắn rằng các cơ sở dữ liệu chỉ ngày càng hoàn thiện mà không bao giờ đạt mức đầy đủ.
Đối với các phân tích chức năng, chúng ta có thể sử dụng các cơ sở dữ liệu sau:
- Databases to identify gene families
- UniRef50
- UniRef90
- Grouping in other functional categories
- MetaCyc Reactions
- KEGG Orthogroups (KOs)
- Pfam domains
- Level-4 enzyme commission (EC) categories
- EggNOG (including COGs)
- Gene Ontology (GO)
- Informative GO
- Pathway reconstruction
Kết quả phân tích
Bảng OTU

Biểu đồ tương tác Krona

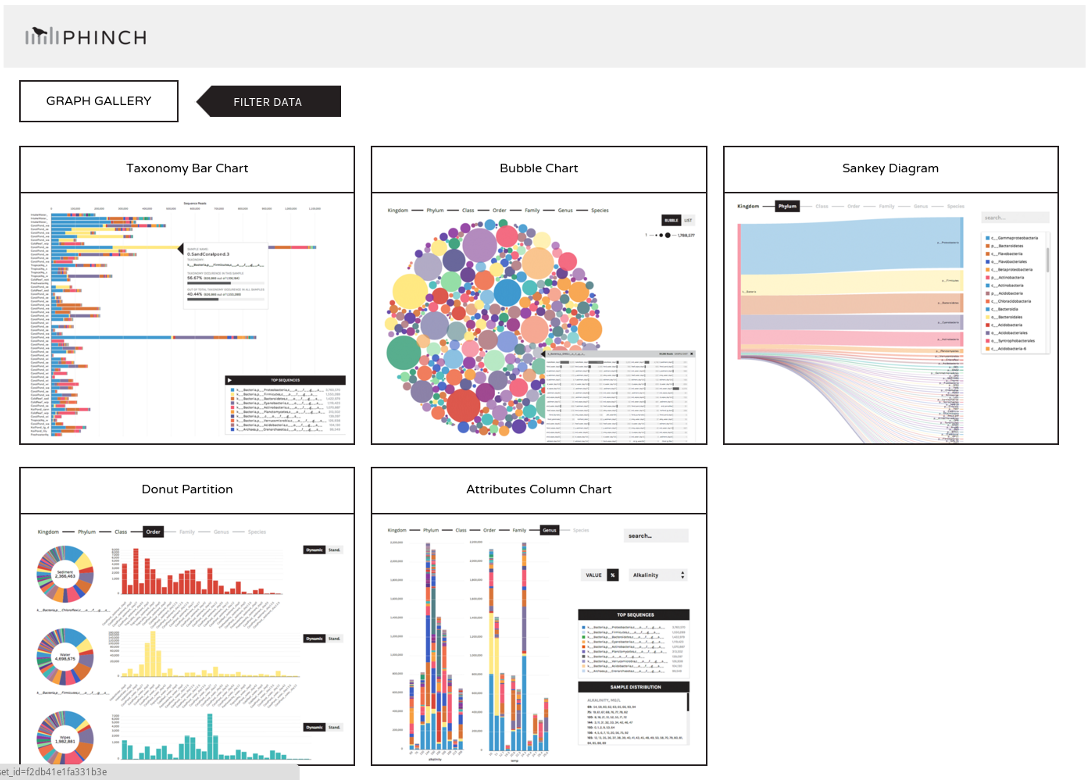
Phinch
Thay lời kết
Metagenomics đã được các nhà nghiên cứu trên thế giới thực hiện nghiên cứu mạnh ngay từ khi các thiết bị đọc trình tự thế hệ mới được thương mại hóa năm 2005 (hình dưới). Cho đến nay (2/2022), đã có khoảng 30,000 nghiên cứu về metagenomics được ghi nhận trên PubMed. Trong khi đó, số nghiên cứu Metagenomics có liên quan tới Vietnam trên PubMed còn rất khiêm tốn.
Hoạt động trong lĩnh vực Đọc trình tự thế hệ mới và Phân tích Tin sinh học, LOBI luôn sẵn sàng tư vấn chiến lược lấy mẫu, kỹ thuật đọc trình tự và quy trình phân tích Tin sinh học mới nhất và tối ưu nhất. Mọi câu hỏi, băn khoăn xin vui lòng liên hệ qua form tại đây, chúng tôi sẽ hỗ trợ bạn.
LOBI Vietnam là công ty tiên phong trong lĩnh vực Đọc trình tự gen thế hệ mới NGS (Next Generation Sequencing) và Phân tích Tin sinh học. Liên hệ hotline/Zalo 092.510.8899 để biết thêm chi tiết.


